
DNA甲基化检测技术介绍
一、应用简介
DNA甲基化是基因表达调控的重要机制之一,DNA甲基化检测是指通过各类技术方法测定肿瘤细胞DNA甲基化程度的检测手段。在恶性肿瘤发展进程中,DNA甲基化状态并非恒定不变:肿瘤细胞全基因组的低甲基化程度与疾病进展、肿瘤大小及恶性程度密切相关。因此,DNA甲基化检测对判断肿瘤恶性程度具有重要参考价值。
二、技术原理
DNA甲基化检测的核心基础是:基于DNA甲基化状态的差异性,通过特异性处理与扩增技术,区分并检测甲基化与非甲基化位点。以下为两种常用检测方法的具体原理:
1. 甲基化特异性PCR(Methylation-specific PCR,MSP)
该方法由Herman等人于1996年在重亚硫酸盐处理技术的基础上建立,具有高敏感性,主要用于甲基化定性研究。核心原理为:采用亚硫酸氢盐处理基因组DNA,使所有未发生甲基化的胞嘧啶(C)转化为尿嘧啶(U),而甲基化的胞嘧啶保持不变;随后分别设计针对甲基化序列和非甲基化序列的特异性引物进行PCR扩增。通过电泳检测MSP扩增产物:若针对处理后甲基化DNA链的引物能扩增出目的片段,说明该检测位点存在甲基化;反之,则说明该位点无甲基化。
特点:适用范围广泛,可精准检测特异位点是否发生甲基化。
2. 亚硫酸氢盐测序法(Bisulfite sequencing PCR,BSP)
该方法由Frommer等人于1992年提出,是研究DNA甲基化的经典方法,具有高可靠性与高精确度,可明确目的片段中每个CpG位点的甲基化状态。核心原理为:用亚硫酸氢盐处理基因组DNA,未甲基化的胞嘧啶转化为尿嘧啶,甲基化胞嘧啶则保持不变;随后设计BSP特异性引物进行PCR扩增,扩增过程中尿嘧啶全部转化为胸腺嘧啶(T);最后通过对PCR产物测序判断CpG位点的甲基化状态,此为BSP-直接测序法。若将PCR产物克隆至载体后再进行测序,可提高测序成功率,该方法称为BSP-克隆测序法。
特点:精确度高,可准确检测出目标区域的甲基化程度百分比。
三、实验方法
1. 核心检测方法
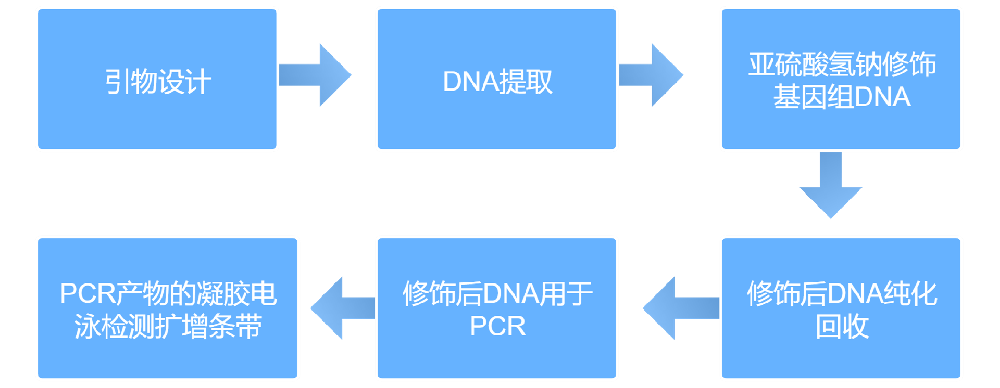
核心环节:DNA甲基化检测(含基因组DNA提取、亚硫酸氢盐处理、特异性引物设计、PCR扩增、产物检测/测序等关键步骤),常用方法包括MSP法、BSP法等。
四、常见问题
1. 如何确定基因的目标区域进行甲基化分析?
转录起始位点附近的CpG岛是DNA甲基化分析的首选区域,该区域通常位于基因的启动子区或5’UTR区(5’非编码区)。
2. 为何文献报导的甲基化序列不在CpG岛中?
不存在明显的CpG岛,并不意味着该区域不会发生甲基化。甲基化也可能发生在CG位点不密集的区域。
3. BSP甲基化扩增得到的PCR产物可否直接测序?为何需要构建TA克隆后测序?
不建议直接对PCR产物测序。若直接测序,可能在原始的C位点出现双峰图(分别对应甲基化的C和未甲基化的T),无法准确判断该位点的甲基化程度;而将PCR产物构建TA克隆后测序,可明确单个克隆的序列信息,进而精准确定甲基化程度。


