
简介
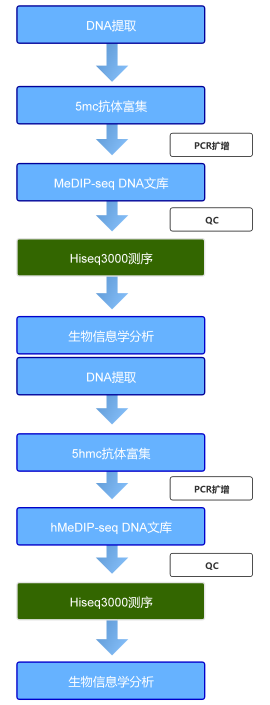
MeDIP-Seq(甲基化 DNA 免疫沉淀测序)是一项基于抗体富集原理的全基因组甲基化检测技术。该技术依托甲基化 DNA 免疫共沉淀技术,借助5'- 甲基胞嘧啶(5mC)特异性抗体,靶向富集基因组中发生甲基化修饰的 DNA 片段,后续结合高通量测序手段,可在全基因组范围内实现对高 CpG 密度甲基化区域的高精度解析。
hMeDIP-Seq(羟甲基化 DNA 免疫沉淀测序)的核心原理与 MeDIP-Seq 完全一致,其区别在于利用5'- 羟甲基胞嘧啶(5hmC)特异性抗体富集基因组中的羟甲基化 DNA 片段。作为一种新发现的表观遗传修饰碱基,5hmC 由 10-11 易位(TET)家族的酶氧化 5mC 生成。该修饰不仅能够削弱 MeCP 蛋白甲基化结合结构域(MBD)与甲基化 DNA 的亲和能力,进而参与基因表达的转录调控,同时还在 DNA 主动去甲基化过程中发挥关键作用。
依托 MeDIP-Seq 与 hMeDIP-Seq 技术,研究人员能够高效、精准地定位基因组中的甲基化及羟甲基化区域,通过对比不同细胞、组织或疾病样本间的 DNA 甲基化修饰图谱差异,为基础生物学机制研究及临床诊疗方案的开发提供坚实的理论依据。
流程图
技术优势
1、全转录组范围研究mRNA的甲基化位点;
2、通过抗体富集的方法,充分利用了抗体的灵敏性;
3、分析方法类似ChIP-seq,数据分析相对成熟。
mRNA甲基化测序
简介
表观遗传修饰并非仅存在于基因组层面(如 DNA 甲基化),在转录组层面同样存在相应的表观修饰类型,且这类修饰承担着关键的转录后调控功能。早在四十年前,研究人员便发现信使 RNA(mRNA)上存在腺嘌呤甲基化修饰,即m?A 修饰。
作为一种广泛存在的 RNA 表观修饰,m?A 的出现频率约为每一条 mRNA 分子上分布 3-5 个修饰残基。该修饰具有可逆性特征,其动态变化过程受到甲基化酶复合体(包含 METTL3、METTL14、WTAP 等核心组分)与去甲基化酶(如 FTO)的精准调控。值得注意的是,m?A 修饰在人类与小鼠的转录组中高度保守,这一特性暗示其可能具有不可或缺的生物学功能。
已有研究证实,m?A 修饰参与调控 mRNA 剪切、生物钟节律维持、mRNA 稳定性维持等多个关键生理过程。除此之外,m?A 还可通过招募 YTHDF2 蛋白,促使处于翻译进程中的 mRNA 从核糖体转移至细胞质加工小体(P-body),并在该区域发生降解;且 mRNA 的降解效率与其携带的 m?A 修饰位点数量呈明显相关性。
在 mRNA 甲基化的检测技术中,甲基化 RNA 免疫沉淀测序(MeRIP-Seq,即 m?A 特异性甲基化 RNA 免疫共沉淀结合二代测序技术) 是目前的主流手段。其核心原理为:哺乳动物 mRNA 的甲基化修饰多发生于腺嘌呤第 6 位氮原子上,基于这一特点,可利用 m?A 特异性抗体靶向富集高甲基化的 mRNA 片段,后续结合第二代高通量测序技术对富集产物进行测序分析,从而实现全转录组范围内 m?A 修饰位点的精准检测。
流程图
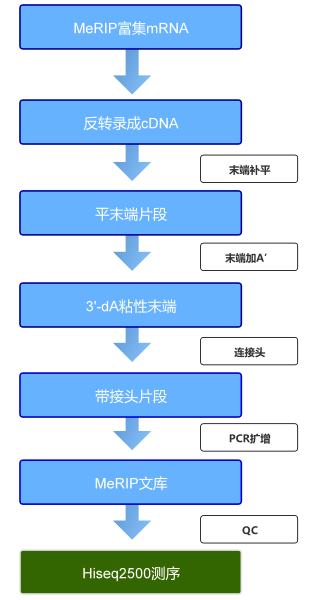
核心技术优势
物种适配性强:可直接对任意物种的转录组高甲基化片段实施测序分析,无需依赖已知的基因组序列信息。
检测覆盖范围广:能够实现全转录组范围内甲基化区域的无差别覆盖,保障甲基化位点筛查的全面性。
定位精准度高:可在目标结合位点上下游 100-200 个碱基的区间内,完成甲基化区域的精准定位。
信号数字化无干扰:直接对甲基化片段进行测序与定量分析,有效规避传统芯片杂交技术中,荧光模拟信号易引发的交叉反应与背景噪音问题。
表观遗传学研究服务注意事项
1. 样本处理与保存
表观遗传修饰状态极易受外界环境因素干扰,样本采集后需即刻开展稳定化处理,例如添加 DNA 甲基化保护剂,同时需杜绝反复冻融操作,防止表观修饰状态发生改变。针对福尔马林固定石蜡包埋(FFPE)样本,需提前对其 DNA 完整性及表观修饰信息的保留程度进行全面评估,确保样本符合实验标准。
2. 技术方案选择
需依据具体研究目的,针对性选择适配技术:开展 DNA 甲基化分析时,可选用全基因组甲基化测序(WGBS)、简化代表性重亚硫酸盐测序(RRBS)等技术;研究组蛋白修饰,则推荐采用染色质免疫沉淀测序(ChIP-seq)技术。需提前与服务提供方确认技术的灵敏度与覆盖度,必要时可整合多组学技术,进一步提升研究的深度与广度。
3. 数据分析与验证
需向服务方明确要求,提供完整的原始实验数据及标准化分析流程,并借助 UCSC 表观基因组浏览器等专业数据库完成数据注释。鉴于表观遗传数据存在显著的个体差异,实验设计阶段需设置足量的生物学重复;同时需通过实时荧光定量 PCR(qPCR)、甲基化特异性 PCR(MSP)等实验手段,对关键研究结果进行验证,确保结论的可靠性。此外,需严格遵守数据保密协议,切实保障样本相关隐私信息的安全。


